ちょっとしたRのこと
クリップボードとのやりとり
クリップボードを経由すれば,他のソフト(例えば Excel や SPSS)との間で簡単にデータを移すことができる。
いちいちオプションを打つのが面倒なので,クリップボードへの読み書きが簡単にできる関数を用意してみる。
(Windows用。Mac は手元にないのでわかりません。)
クリップボードから読み取ってデータフレームをつくる関数
read.cb <- function(header=TRUE, ...)
utils::read.table(file="clipboard", header=header, ...)
クリップボードにデータフレームを書き込む関数
write.cb <- function(data, sep="\t", header=TRUE, row.names=FALSE,
col.names=ifelse(header && row.names, NA, header), qmethod="double", ...)
utils::write.table(data, file="clipboard", sep=sep, row.names=row.names,
col.names=col.names, qmethod=qmethod, ...)
この2つを .Rprofile に書いておけば幸せになれる。
ver. 2.5.0 にて read.table(), write.table() は base パッケージから utils パッケージへ変更になりました。
用法
Excel から読み込むときは,R に渡す範囲を(変数名を含む行つきで)選択->コピーしてから,Rで
read.cb()->my.data
とでもすればよい。変数名を用意していない場合ならば
read.cb(h=F)->my.data
逆に,R から Excel にデータフレームを渡すときは,
write.cb(my.data)
としてから Excel 上で貼り付けすればOK。
これらの引数のデフォルト値は Excel とのやり取りを想定している。
Excel 以外のソフトとのやり取りをメインにしている人はデフォルト値を適宜
書き換えて使うほうが便利。
foreign::read.spss() を使えば,SPSS データファイルからの読み込みができるし,
package RODBC や package gdata を使えば Excel スプレッドシートを読み込めます。
しかし,シート内容がデータだけで構成されていなかったりして,グリッドの一部分を取り出して読み込みたいときは,
クリップボードを使うのが楽。
スクリプトの読み込み
メニューの "Source R code..." (日本語メニューだと「R コードのソースを読み込み...」)は,
日本語版Windowsでの日本語のファイルパスを正常に処理してくれないので困る(現在v2.3.0でもダメ,XPでしか確認してません)。
手でパスを打つのもD&Dも面倒くさいので,こんな関数を書いてみる。
日本語ファイルパスの問題はv2.4.0で解消されました。カスタマイズのヒントになるかもしれないので以下の関数は置いておきます。
src <- function(file=file.choose(), chdir=TRUE, ...)
base::source(file=file, chdir=chdir, ...)
これで
src()
とやれば,「R コードのソースを読み込み...」と同様のダイアログが開いてファイルを選択できる。
日本語パスの扱いも正常。
また,上記関数は他の引数のデフォルト値を chdir=TRUE にしてあるので,
スクリプト内でのデータファイルのパスなどをスクリプトからの相対パスで書くことができる。この辺はお好みで。
CRANのミラーサーバ
パッケージ更新などの接続先サーバを日本のものにセットしたければ,
options(repos=local({ r <- getOption("repos"); r["CRAN"] <- "http://cran.md.tsukuba.ac.jp/"; r }))
この例では筑波大学のミラーサーバなので,他のところにしたければ URL の部分を書き換え。
| 会津大学 | ftp://ftp.u-aizu.ac.jp/pub/lang/R/CRAN/ |
| 兵庫教育大学 | http://essrc.hyogo-u.ac.jp/cran/ |
| 筑波大学 | http://cran.md.tsukuba.ac.jp/ |
| 東京大学 | ftp://ftp.ecc.u-tokyo.ac.jp/CRAN/ |
| 統計数理研究所 | http://cran.ism.ac.jp/ |
| 岡田さん個人運営サーバ | http://cran.okada.jp.org/ |
でもね,実際のところ,日本のサーバよりも西海岸のサーバにつないだ方が軽いときもあったりする。:-(
このCRANを指定するオプションはバージョン2.1.0で変更されたものなので注意。2.1.0より前のバージョンを使っているなら,
options(CRAN="http://cran.md.tsukuba.ac.jp/")
ヘルプ表示
デフォルトで
help(par)
などと打って現れるヘルプは,R GUI の別ウィンドウで表示され,この別ウィンドウは内部のテキストの検索もできない
(他にコピペなどする必要がある)不便なものである。そこで,
options(htmlhelp = TRUE)
としておけば,help() で表示されるヘルプがブラウザで開くようになる(別オプションでブラウザの指定も可能)。
これで検索も楽々。
Windowsユーザならば,
options(chmhelp = TRUE)
とすれば,コンパイル済みHTMLヘルプで表示できる。目次やキーワード検索も使えて便利。
v2.4.0からは,インストールウィザードにてデフォルトのヘルプ形式をこれらから選択できるようになりました(Rprofile.site を書き換えている)。
v2.10.0から,ローカルで R がヘルプ用のhttpサーバを立ち上げてブラウザで表示する方式になりました。インストール時に従来のものとの選択はできるようです。
ライブラリ パス
開発された当初からだと思いますが,パッケージの探索場所(ライブラリ)は設定で変更できます。ところが,ver. 2.5.x あたりからパス設定の種類を増やされて,何が何だかわかりづらい。
よって,ここに簡単なまとめを載せておきます。
ちゃんと用語を区別して使いましょう。Rでは「パッケージ」を置いておく場所(ディレクトリ)が「ライブラリ」です。
base パッケージ の変数
| 名前 | デフォルト値 | 説明 |
| .Library | ${R_HOME}/library | 基本。インストーラを使うと推奨パッケージがインストールされる場所。 |
| .Library.site | ${R_HOME}/site-library | v2.5.0から。普通に使っていると ${R_HOME}/site-library が存在しないので character(0) です。baseパッケージの変数ですが,Rprofile.site内にコードを書くことで値をセットできます。 |
環境変数
| 名前 | 説明 |
| R_LIBS | 設定されると,.libPaths() の最初に挿入されます。Windowsの場合,リストはセミコロン区切り |
| R_LIBS_USER | v2.5.0から。未設定だと,${R_USER_HOME}/R/win-library/x.yで初期化されます(付属ドキュメントは誤記です。v2.8.0)。x.y は R のバージョン番号 x.y.z の数字。{R_USER_HOME} の決定され方は下記。
パスに特定の文字列を埋め込むことで,バージョン番号などの展開ができます |
| R_LIBS_SITE | 設定されていると,起動時に .Library.site の初期化に使われます。同様に,Windowsではセミコロン区切り。
R_LIBS_USER と同様に,パスに特殊な展開文字列を使用できます |
library() や require() は .libPaths() に列挙されているディレクトリを探します。install.packages() などは,lib 引数を省略するとデフォルトでは .libPaths()[1] を使います。
.libPaths() の返す値は,起動時にデフォルトで R_LIBS, R_LIBS_USER, .Library.site, .Library の順のリスト(のうちディレクトリが存在しているもの)で初期化されます。
.libPaths(new) を使ってライブラリパスをセットした場合,.libPaths() の返す値は,unique(c(new, .Library.site, .Library)) のうちディレクトリが存在しているパスのベクトルになります。
上記 ${R_USER_HOME} (ドキュメントでは home directory と呼ばれる) は次の順で決定されます:
- 環境変数 R_USER が設定されている場合,その値
- 環境変数 HOME が設定されている場合,その値
- ユーザーの "personal" ディレクトリ(いわゆるマイドキュメント)
- 何らかの理由で "personal" ディレクトリが存在しない場合,2つの環境変数 ${HOMEDRIVE}${HOMEPATH} をつなげた値。これは通常はマイドキュメントの一つ上のディレクトリ。
R だけでパス図を描く
構造方程式モデリング (SEM) と言えばパス図 (path diagram),パス図といえば SEM 。
R でも古くから sem パッケージがあるけど,
そこで指定したモデルのパス図を描くには別途 GraphViz のインストールが必要だった。
そんな折り,Google さんが Chart API に GraphViz を導入してくれたので,これを利用して簡単に R だけでパス図を表示できるよう,スクリプトを書いてみた。
plot.sem.R (GPL2)
使い方は非常に簡単:
source("plot.sem.R")
library(sem)
# データとモデルの例は help("sem") から
R.DHP <- read.moments(diag=FALSE, names=c("ROccAsp", "REdAsp", "FOccAsp",
"FEdAsp", "RParAsp", "RIQ", "RSES", "FSES", "FIQ", "FParAsp"))
.6247
.3269 .3669
.4216 .3275 .6404
.2137 .2742 .1124 .0839
.4105 .4043 .2903 .2598 .1839
.3240 .4047 .3054 .2786 .0489 .2220
.2930 .2407 .4105 .3607 .0186 .1861 .2707
.2995 .2863 .5191 .5007 .0782 .3355 .2302 .2950
.0760 .0702 .2784 .1988 .1147 .1021 .0931 -.0438 .2087
model.dhp <- specify.model()
RParAsp -> RGenAsp, gam11, NA
RIQ -> RGenAsp, gam12, NA
RSES -> RGenAsp, gam13, NA
FSES -> RGenAsp, gam14, NA
RSES -> FGenAsp, gam23, NA
FSES -> FGenAsp, gam24, NA
FIQ -> FGenAsp, gam25, NA
FParAsp -> FGenAsp, gam26, NA
FGenAsp -> RGenAsp, beta12, NA
RGenAsp -> FGenAsp, beta21, NA
RGenAsp -> ROccAsp, NA, 1
RGenAsp -> REdAsp, lam21, NA
FGenAsp -> FOccAsp, NA, 1
FGenAsp -> FEdAsp, lam42, NA
RGenAsp <-> RGenAsp, ps11, NA
FGenAsp <-> FGenAsp, ps22, NA
RGenAsp <-> FGenAsp, ps12, NA
ROccAsp <-> ROccAsp, theta1, NA
REdAsp <-> REdAsp, theta2, NA
FOccAsp <-> FOccAsp, theta3, NA
FEdAsp <-> FEdAsp, theta4, NA
sem.dhp.1 <- sem(model.dhp, R.DHP, 329,
fixed.x=c('RParAsp', 'RIQ', 'RSES', 'FSES', 'FIQ', 'FParAsp'))
plot(sem.dhp.1) # !!! これ !!!
するとグラフィックウィンドウが開いて,パス図の画像が表示される。
plot() には sem::path.diagram() の引数がそのまま使えるので,図の調整もできる。たとえばこんな感じ:
plot(sem.dhp.1,
min.rank='RIQ, RSES, RParAsp, FParAsp, FSES, FIQ',
same.rank='RGenAsp, FGenAsp',
max.rank='ROccAsp, REdAsp, FEdAsp, FOccAsp',
rank.direction="TB", edge.labels="both", standardize=TRUE)
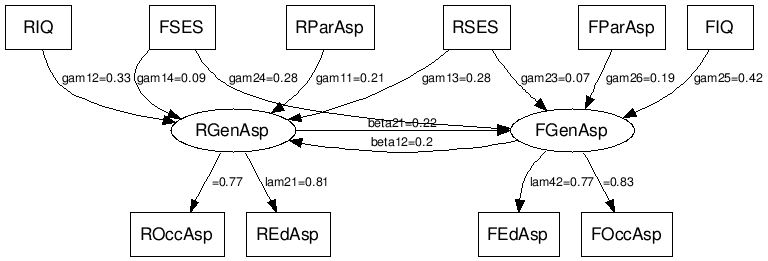
このスクリプトは画像表示に rgdal パッケージを使っているので,sem だけでなくこちらも前もって入れておく必要がある。
だが,plot 引数を FALSE にすれば画像ファイルは生成されても表示をしない。
この場合 rgdal パッケージが入ってなくてもエラーにならないので,とりあえず rgdal 導入前にパス図の具合を見てみたい人は
plot(sem.dhp.1,
min.rank='RIQ, RSES, RParAsp, FParAsp, FSES, FIQ',
same.rank='RGenAsp, FGenAsp',
max.rank='ROccAsp, REdAsp, FEdAsp, FOccAsp',
rank.direction="TB", edge.labels="both", standardize=TRUE,
plot=FALSE, file="test.png")
などとして,出てきた PNG ファイルを開いてみればよい。file を指定しないとテンポラリファイルを使うため自動的に削除されるので注意。
SEM を使う場面ではモデルの試行錯誤が多いと思うが,これはそんなときに簡単にパス図でモデルを確認するために作ったもの。
画像もウェブ用なので荒い。
いざ最終成果を出版するときは,自分のマシンに GraphViz を入れて SVG や PDF を生成することをお勧めする。
ついでに,sem パッケージでは sem() が成功しないとパス図が書けない。
それでは試行錯誤に不便なので,モデル指定だけでパス図が書けるように path.diagram() の拡張を書いてみた:
path.diagram.R (GPL2)
ANOVA4 形式のデータをRで扱う
広島女学院大学 桐木建始さんのANOVA4用のデータ形式(ANV)をRで扱えるように(リソースの再利用),関数を書きました。
anova4.R (GPL2)
自由に利用してください。(R への移行と学習のために)
これでもう ANOVA4 on the Web は必要ない,というか,
ウェブ上でもそれ以上のことが Rweb gateway でできる。
関数概説
read.anv-
ANVファイルからデータを読み込みます。
返るのは read.table() などと同じくデータフレームです。
読み込むだけなので,その後分散分析に限らずいろいろな分析を適用できます。
また,'anv4' という特殊なクラスが付いています。これがANV形式データのしるしです。
以下の関数を使うときに単なるデータフレームでなくこのクラスであることが必要になります。
write.anv'anv4' クラスのオブジェクトをANVファイル形式にしてファイル(もしくはコネクション)に書き出します。
引数はANV形式テキストへ変換が可能な 'anv4' クラスが付いたデータフレームでなければなりません。
as.anv4'anv4' クラスのオブジェクトを作ります。通常のデータフレームなどから
'anv4' クラスを持つデータフレームを作ることができます。
これを利用することで,write.anv() と組み合わせて通常のデータフレームをANV形式ファイルに出力したり,
通常のデータフレームに anv4.fit() を適用したりすることができます。
anv4.fit'anv4'クラスのデータに分散分析モデルをフィットさせます。
基本として多層の stats::aov() を適用します。
加えて,stats::lm() や nlme::lme() を適用します(可能ならば)。
これらのフィット関数の返り値をリストしたものが返ります。
このリストには 'anv4.fit' というクラスが付いています。
stats::lm() は,固定効果しか分析できません。
これを追加で適用している意味は,場合によって異なります。
モデルがブロック内因子を含んでいる場合,stats::lm() の結果は
ブロックを固定因子を見なした場合の結果を示しています
(最初の stats::aov() の適用ではブロックを変量因子と見なしています)。
モデルがブロック内因子を含んでいない場合,stats::lm() の結果は
単に stats::aov() ではなく stats::lm() も使ってみた(戻り値のクラスが異なる)というくらいで,
さほど重要な意味はありません。
nlme::lme() の結果も,nlme::lme() を使ってみた(戻り値のクラスが異なる)というくらいで,
さほど重要な意味はありません。おそらく stats::aov() と同じ数値が得られるでしょう。ただし,
R の関数のいくつかは引数に取れるオブジェクトのクラスを限定しますから,クラスが異なることは場合によっては重要です。
※ 以下の関数は忘れて結構。
print.anv4.fit'anv4.fit' 用の print() です。
現在の仕様では,stats::aov() のフィット結果だけを print() します。
anova.anv4.fit'anv4.fit' 用の anova() です。
'anv4.fit' リストに含まれるすべてのフィット結果について分散分析表を書きます。
使用例
sample_data <- read.anv("http://ofmind.net/doc/r-tips/sample.anv")
sample_fit <- anv4.fit(sample_data)
anova(sample_fit)
EmEditor用
リクエストに応じて,EmEditor 用の R 言語構文ファイルを出力する R スクリプトを書きました。
(Rweb gateway からの転用です。EmEditor は設定に限界があるのでハイライトだけなら Rweb gateway のほうが高性能!?)
emeditor_syntax_file.R (GPL2)
読み込んで,
emeditor_syntax_file("my-R.esy")
とやると構文ファイルを出力してくれます。
キーワード同定の正確さを上げるために,正規表現を使用しています。
これが,マシンの性能によっては辛いかもしれません(EmEditorが幾分重くなります)。
そんなときは,
emeditor_syntax_file("my-R.esy", FALSE)
とやると,正規表現を使わない版を出力してくれます。こちらなら大抵のマシンで大丈夫と思いますが,正確さを犠牲にしているので
my.data <- rnorm(100)
などが data の部分だけハイライトされてしまいます。妥協してください。
この関数は,関数を実行する R の環境によって出力内容を変えます。
例えば,何か追加パッケージをロードしている状態だとそれが強調キーワードに反映されます。
自分で変数を作っているなら,それも強調キーワードに含まれます。
よって,デフォルトな構文ファイルがほしいなら起動直後の状態で関数を実行してください。
マクロで実行
ついでにマクロ作ってみました。選択範囲のスクリプトを R で実行して結果を EmEditor のアウトプットバーに出します。
RunR.jsee (GPL2)
使い勝手を良くするにはこれだけでなくもう一工夫いるので,参考までに。
検索バー で R Site Search
Firefox 検索バー用の OpenSearch プラグインです。RSiteSearchアドオンがまだ Firefox 3 に対応していないから,あるいは,アドオンよりも検索バーを好むから,という方はお使いください。
RSiteSearch.xml
ファイルをローカルに保存して,下記のどちらかに入れて Firefox を再起動すると有効になります。
- <Firefoxをインストールしたディレクトリ>/searchplugins (システム全体)
- Windows XP では一般的には C:\Program Files\Mozilla Firefox\searchplugins
- <Firefoxプロファイルのディレクトリ>/searchplugins (ユーザごと)
- Windows XP では一般的には C:\Documents and Settings\<ユーザ名>\Application Data\Mozilla\Firefox\Profiles\<プロファイルコード>\searchplugins
あるいは,現代的ブラウザなら,ここをクリックすればインストールできます。
OpenSearch プラグインは Internet Explorer 7 でも使えるらしい。
R User Configurationについて
思ったより需要が多いようなので,ここに多少説明を載せておきます。
ダウンロードもここからどうぞ。
R User Configuration version 0.11 (47,861 Bytes) GNU GPL 2
ライセンスを変更しました。ご注意ください。
R の version 2.8.1 にて動作確認しました。古い R では現在の R User Configuration はうまく動かないので,
R User Configurationの昔のバージョンが必要な人は連絡してください。
概要
R User Configuration は,Windows NT系の最近のOS (2000/XP/Vista) にインストールした R のユーザ個別設定を行うものです。
最初にこれを自分たち用に開発した動機は,Windows版 R の公式のインストーラが不親切だったからです(これを書いている今もです; v2.8.0)。
他のOS用のも不親切なのかどうかは知りませんので,Macや*nix系への対応は端から念頭にありません。
Windows 9x系は対応してもよいですが,テスト環境を作ったり大幅な改変をしたりする必要があり,大変なので放置しています。
試していませんが,9x系でうまく動いたならラッキーと思ってください。
当初,このツールは日本語用設定を行うことを目的に開発されました。受講者に設定ファイルの編集を説明するのが面倒だったからです。
その後,R利用上の不満を解消するための機能をいくつか追加しました。
また,Rの様々な活用法を探索するためのテスト実行補助ツールとしても使用できるようにしました。
ファイル内容
ダウンロードしたアーカイブファイルを展開して出てくるのは, "R user config.hta" というファイル1つだけです。
これで正しいです。通信エラーがあったり私がzipするときに間違えたりしている訳ではありません。
hta ファイルにすべてが含まれています。
使い方
- ダウンロードした zip ファイルを展開する
- 出てきた R user config (.hta) をダブルクリックする
- 現れた画面の右側にある「オート」欄のボタン(設定しておくれボタン)をクリックする(下図の赤枠)
- これでおしまい! 不幸にもエラーダイアログが出ちゃった場合は自分でなんとかするか私に相談するかしてください
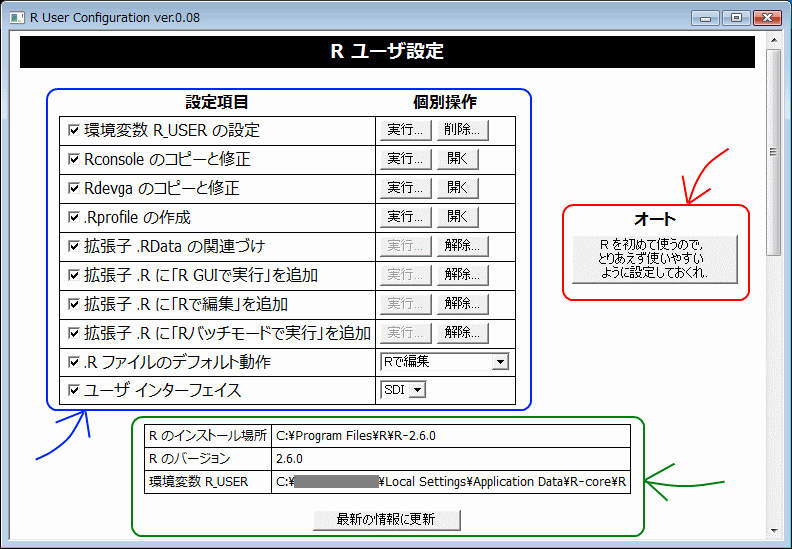
上は version 0.08 の図です,ご勘弁。
カスタマイズ好きの使い方
- 画面の左側にある表(上図の青枠)を見ながら,項目個別に設定する
- 設定したい項目の「実行」ボタンを押す
- 設定解除したい項目のボタン(「削除」,「解除」)を押す
- 編集したいファイルの「開く」ボタンを押す(メモ帳が起動します)
- 変更したい項目のドロップダウンリストを選択する
-
画面下部にある表(上図の緑枠)は,それぞれ下記項目の現在状態です。
- Rがインストールされている場所
- インストールされているRのバージョン
- R_USERに設定されているディレクトリ
R User Configurationを起動した後にインストールしたり設定変更したりした場合は「最新の情報に更新」ボタンを押すとこのツールに反映されます。
複数のバージョンの R を1つのマシンにインストールした場合は,後からインストールしたもので情報が上書きされますので,
「Rのインストール場所」「Rのバージョン」の項目については複数インストールされている R のうちバージョンが最新のものを反映しているという保証はありません。
Rマニアな使い方
画面を下にスクロールして,中級者向けのところを見てください。それなりにレイアウトしていますので,それぞれが何をするものかは見た感じでわかると思います。
逆に,見てわからないなら手を出さない方が無難です。
注意事項
- R のインストーラを実行したときに,「バージョン番号をレジストリに保存する」のチェックを外さないようにしてください。
- いくつかの設定項目ではレジストリを書き換えるため,それらの機能を利用するなら(オートも含む),Windows Vistaでは管理者権限でこのツールを実行する必要があります。
更新履歴など
ファイル(.hta)をエディタで開いてください。中に書いてあります。
技術的補足
HTAな理由
R User Configuration は HTA (HTMLアプリケーション) です。
この実装方法を採用している理由は,第一に,この設定ソフトが裏で何をやっているかを利用者が完全に把握できるようにするためです。
ただでさえこんな怪しげな一個人が作成している怪しげなアプリケーションなのですから,下手をすると自分のWindowsを使用不能にされるかもしれないのに,
何をやっているかがわからないものを実行したくありません。
R User Configuration は HTA であり,完全なオープンソースです。
HTA を採用している第二の理由は,現在シェアの大部分を占めるOSにて,何の下準備もインストールもせずにダブルクリックだけでこのツールが動いてほしいからです。
誰だって面倒な手間はかけたくありませんし,欲しいもの(この場合 R の設定)がすぐ手に入るほうがうれしいはずです。
幸いにして最近の Windows は標準で HTA を実行できるようになっていました。
ただ,HTA でできる範囲には限りがありますので,ユーザ用設定にこの範囲を超える機能が必要ならば他の実装方法に乗り換えることも考えるでしょう。が,現在のところ HTA で間に合っています。
第三に,HTA ならテキストエディタだけで開発できます。これはとてもすばらしいことです。あなたはメモ帳ひとつでこのツールの動作や見た目を変更できます。
シェル拡張
いつ頃からかRスクリプトファイルのコンテキストメニューに項目を追加する機能を盛り込みましたが,これはクリップボード+sendkeysという非常に強引な実装をしています。
よって,この機能がある程度予想通りに動作するには,実行時のタイミング,PCの処理性能,ユーザが他の操作しないこと,などいろいろな要因が絡んでいます。
あんまり期待せず,実験的なものだと思って下さい。動作しなくても文句つけないでください。
こうなっているのは,HTA & WSH だけで実装しようという制約から来ています。
他の方法を使えばもっとまともな実装ができますが,今のところ行っていません。要望がたくさん来れば対応するかもしれません。
Rgui.exeが多重起動チェックとか渡されたファイルをエディタでオープンなどのコマンドラインオプションを実装すれば済む話だし,
そっちのほうが周辺ソフトでごちゃごちゃやるよりもまともな方向だと思うので,放置している,という事情もあります。
Windows Vista
Vistaでは,悪名高いUACだけではなく,ファイルシステムまわりも変更されたので,Rもその動作に影響を受けています。
unlink()関数がjunctionに対応していないので,おかげでこれを用いて書き込み可能チェックを行っている
install.packages()関数などもまともに動作しません。
これに対処するために,「オート」ではおそらくjunctionを経由しないであろうパスをR_USERに設定することにしました。
しかし,これも完全な解決ではありません。プリインストールで使っている場合はたいていjunctionを回避できるかと思いますが,
どんなVistaでもこのパスがjunctionにすり替えられていないなどとは言えませんし,そもそも%LocalAppData%は変更できます。
ですから,一般ユーザは R が Vista をフルサポートしてくれるのを待つしかありません。
もともと,互換性のためにLocal Settingsなど昔ながらのパスをわざわざjunctionで作ってあるのですが,Rは救われなかったようです。

